近日,我校物质学院李健课题组开发出基于丝氨酸整合酶(serine integrase)的合成生物学新技术,相关成果在线发表于国际知名学术期刊《核酸研究》(Nucleic Acids Research)。
丝氨酸整合酶通常来源于噬菌体。在噬菌体侵染宿主细胞的过程中,丝氨酸整合酶催化DNA之间发生位点特异性重组反应,促使噬菌体DNA整合至宿主的基因组内。近年来,丝氨酸整合酶被广泛运用于基因组编辑、线性DNA重组、基因线路设计等合成生物学领域。李健组借用生态学领域“共生”的概念,将丝氨酸整合酶、基因线路设计与合成生物学元件等紧密结合,在体内和体外两种应用场景下,实现了对启动子、终止子和表达基因等合成生物学相关元件的精准操控,为合成生物学领域提供了一种基于丝氨酸整合酶的新型遗传操作手段。
研究人员设计了一种“锁-钥契合”模型,将启动子、终止子、丝氨酸整合酶识别位点等合成生物学元件进行排列组合,开发出“锁”模块,将丝氨酸整合酶视为“钥匙”模块,当“锁”与“钥”正确配合即可“开锁”,基因线路便精准开启,并实现不同色素蛋白的表达。此模型用少数的丝氨酸整合酶即可控制多种不同的大肠杆菌实现信号输出,成功实现“输入信号”与“输出信号”之间的指数级增长(图1);而且通过设计“二进制-十进制”信号转换器,也能使同一种大肠杆菌处于多种不同的状态(图2)。此外,研究人员利用“资源分配器”基因线路,对“锁-钥契合”模型进行改进优化后,可使“输出信号”得到显著放大。
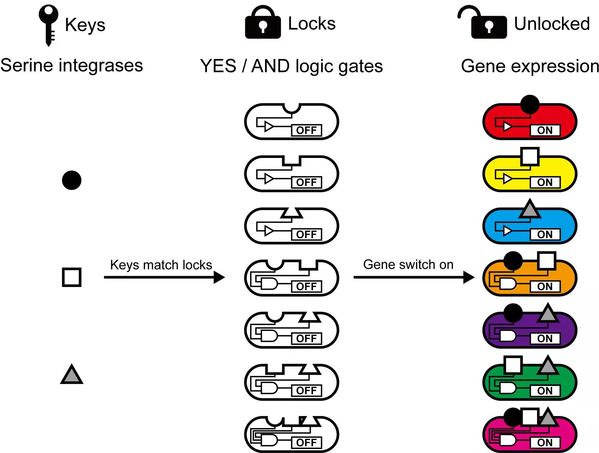
图1 基于丝氨酸整合酶设计的“锁-钥契合”模型,实现信号指数级(y=2^x-1)增长
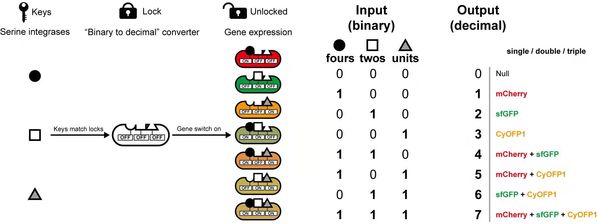
图2 基于丝氨酸整合酶设计的“锁-钥契合”模型,实现“二进制-十进制”信号转换
研究人员还开发出了体外构建融合质粒的新技术,利用丝氨酸整合酶体外催化带有不同识别位点的“供体/受体”质粒,通过抗性筛选方式即可快速获得“供体-受体”融合质粒,并能实现多轮质粒融合及质粒叠加(图3)。值得指出的是,这里的质粒构建方式是以两个环状质粒为直接模板,避免了传统方法中需先制备线性片段再构建质粒等繁琐的遗传操作过程。
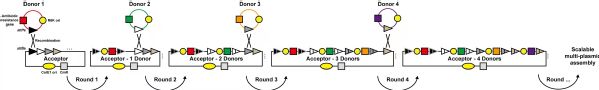
图3 利用丝氨酸整合酶实现体外多轮质粒融合及叠加
研究人员利用质粒融合技术,将6种能够表达不同色素蛋白的质粒进行两两或三三融合,共构建了35种不同表达质粒。再将这些质粒转入大肠杆菌进行蛋白表达,进而获得了一系列带有不同颜色的大肠杆菌(图4),其中一些新产生的颜色是此前在大肠杆菌中从未报道过的。此外,研究人员还提出了一套颜色的标准化流程,利用比色卡,将肉眼可见的颜色转换为标准化的RGB数值。这些彩色大肠杆菌可用于不同的应用场景,比如作为活体绘画的颜料、信息加密的载体及环境响应的媒介等等;同时,这些彩色大肠杆菌的构建策略还具有用于构建其它彩色微生物的可能,进而丰富彩色蛋白/微生物的应用范围。
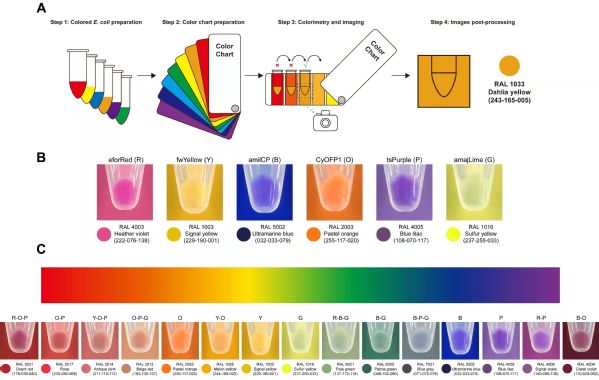
图4 大肠杆菌中不同融合质粒表达色素蛋白。(A)菌体颜色标准化流程;(B)六种基础色素蛋白颜色;(C)能够覆盖完整色谱的代表性彩色菌体展示
上海科技大学物质科学与技术学院2021级博士研究生巴方为论文第一作者,李健教授为通讯作者,上海科技大学为唯一完成单位。
论文标题:SYMBIOSIS: synthetic manipulable biobricks via orthogonal serine integrase systems


 沪公网安备 31011502006855号
沪公网安备 31011502006855号