基于CRISPR的基因编辑工具在疾病治疗等领域拥有重要的应用前景。目前广泛使用的Cas9和Cas12a因分子尺寸较大(超过1000个氨基酸)难以被腺相关病毒(AAV)载体高效递送,限制了其的应用。为此,研究人员积极寻找更小的CRISPR效应核酸酶,其中最紧凑的一类微型核酸酶Cas12f因其较小的分子量(400-700个氨基酸)和较高的编辑活性备受关注。然而大多数Cas12f1仅识别富T的PAM序列,其靶向范围有限,亟需开发新的高活性且靶向范围更广的CRISPR-Cas12f系统。Clostridium novyi Cas12f1 (CnCas12f1,497氨基酸) 对罕见的富C PAM序列具有识别特异性,但只在试管中检测到DNA切割活性,并未发现其具有活细菌内的靶向干扰活性,且其富C PAM的特异性识别和DNA催化切割机制亦不清晰。
2023年9月11日,上海科技大学物质科学与技术学院季泉江教授团队在学术期刊Nature Chemical Biology上发表最新研究成果,解析了微型基因编辑系统CRISPR-CnCas12f1的三元复合体结构,揭示了其独特的结构特征与识别富C PAM并发挥催化作用的分子机制,并通过工程化改造引导RNA,将最初没有编辑活性的CnCas12f1转化为人类细胞中高效的基因编辑工具。

研究人员利用冷冻电镜技术发现,两个CnCas12f1单体以不对称二聚体的形式与引导RNA和目标DNA相互作用,形成CnCas12f1-sgRNA-dsDNA三元复合体。通过层层解析CnCas12f1二聚体的交互界面,进一步明确了形成二聚体的关键作用力:底层主要由范德华力推动,中间两层则主要通过氢键形成,而顶层则依赖于REC识别结构域的α3螺旋间相互作用实现二聚化。
本工作还阐明了CnCas12f1与靶向DNA的相互作用机制,揭示了CnCas12f1通过其识别域REC.1、楔形域WED.1和REC.2来协同识别特定的PAM序列,并通过点突变实验证实了其中5个关键氨基酸残基的重要性。同时,研究人员使用工程化CnCas12f1的“共价二聚体”进行体外切割实验,确定了CnCas12f1的DNA催化切割主要依赖RuvC.1域的活性中心,为优化Cas12f1的基因编辑应用提供了依据。
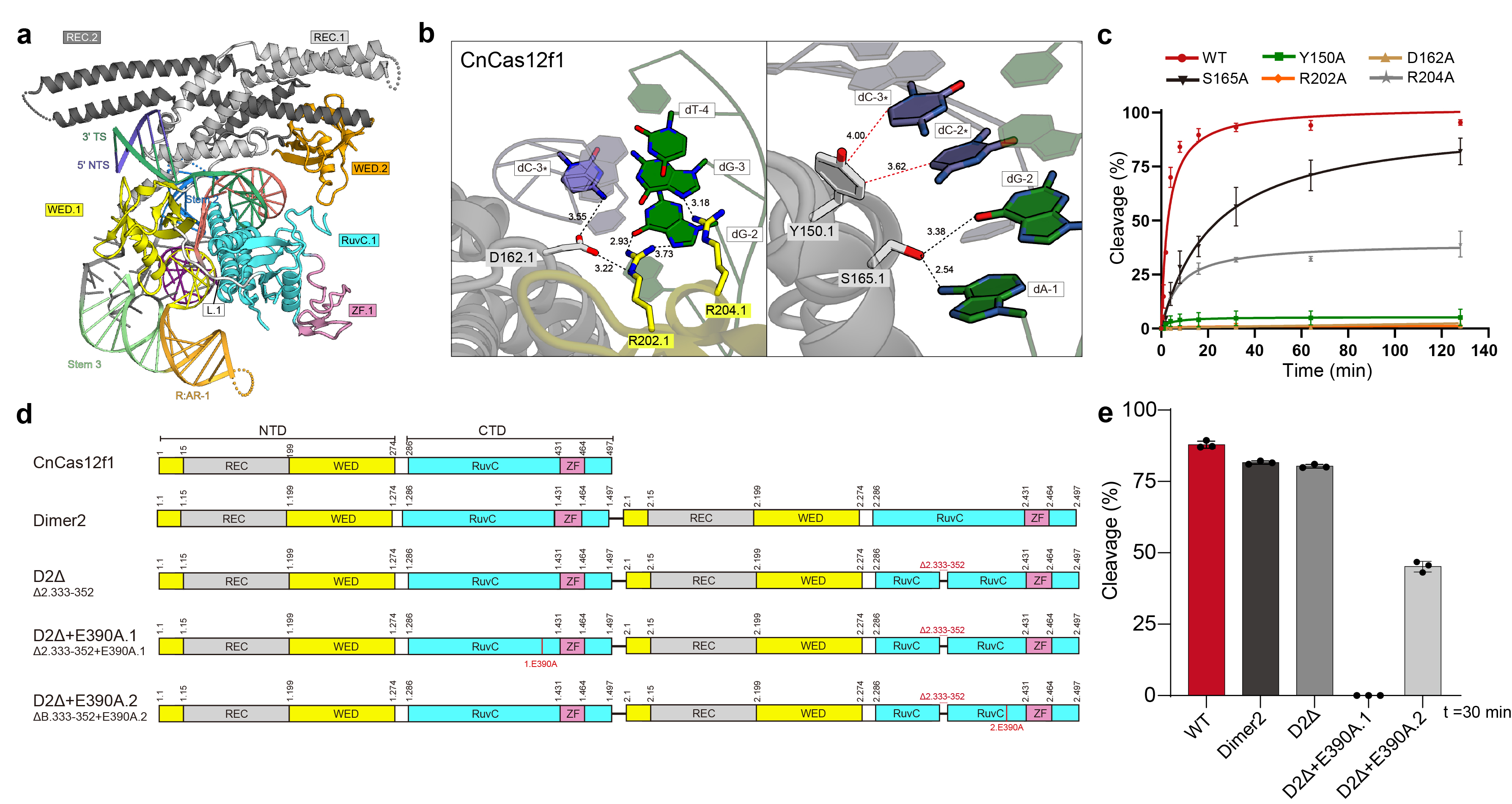
图1:CnCas12f1识别和切割DNA的分子催化机制
系统表征结果证明,CnCas12f1是一个依赖Mg2+且盐浓度敏感的嗜热核酸酶,能够与sgRNA结合后识别并切割含有5'-NCCD (D表示A,G和T) PAM的双链DNA,在靶向链上引入一个切口,非靶向链上引入两个切口,形成不对称粘性末端。
为了获得体内活性,研究人员通过优化引导RNA的结构,设计了一个在细菌内具有高活性的单向导RNA(sgRNA_MS1)。进一步对CnCas12f1的sgRNA进行系统工程化设计,最终获得了在人类细胞中表达更高、编辑活性大幅提升的sgRNA_MS13。这同时也表明引导RNA的结构缺陷是限制Cas12f家族核酸酶基因组编辑活性的主要因素。
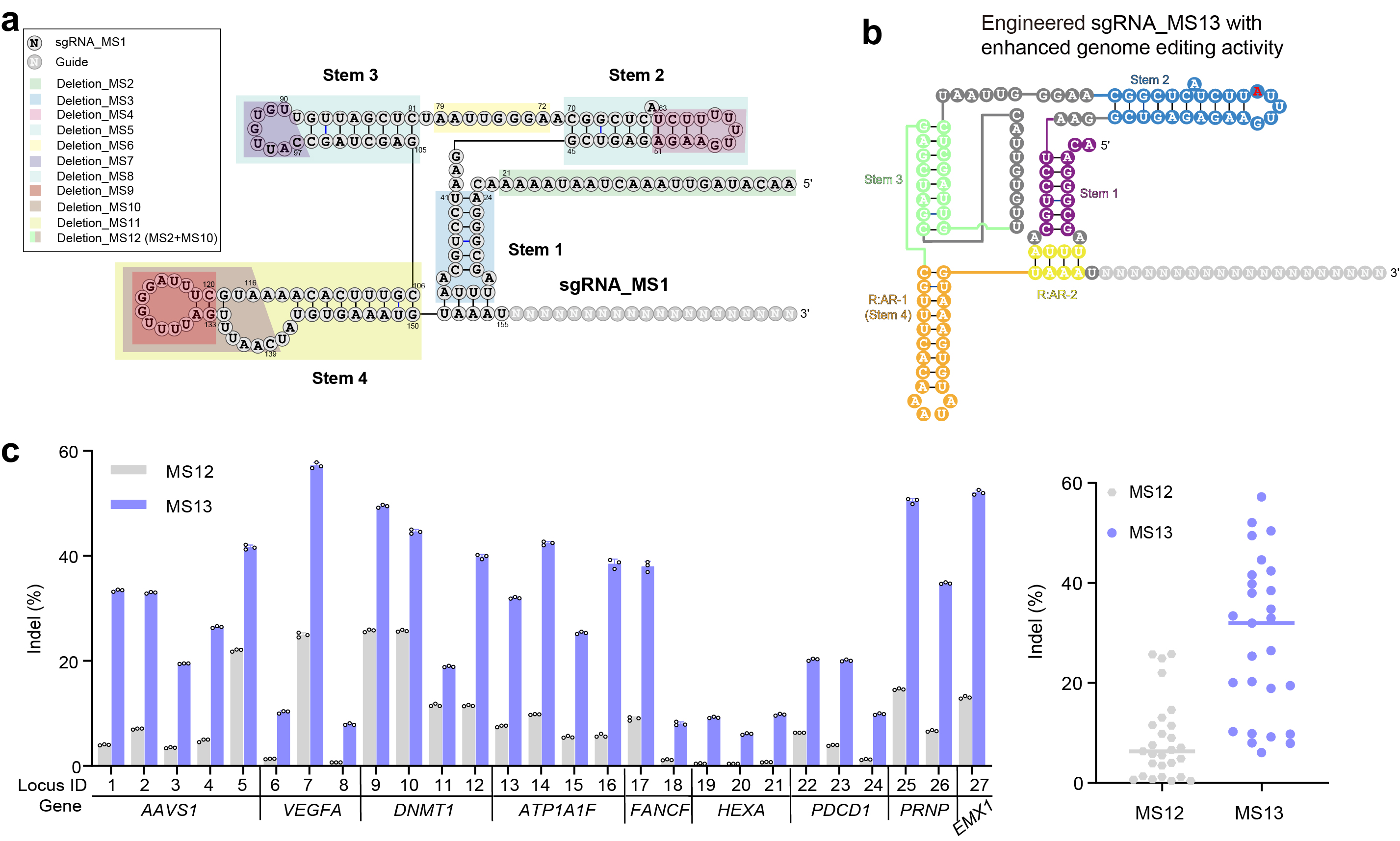
图2:引导RNA工程化提高CnCas12f1的基因组编辑活性
本工作又一次拓展了微型CRISPR核酸酶工具库,增进了对CRISPR-Cas12f家族的结构生物学理解,为开发适合病毒载体递送的高效微型CRISPR系统提供了重要参考。
上海科技大学季泉江课题组博士生苏梦娇、博士后李帆和博士后王玉珏为共同第一作者,季泉江教授与助理研究员吴兆韡为共同通讯作者。上海科技大学为第一完成单位。
文章标题:Molecular basis and engineering of miniature Cas12f with C-rich PAM specificity


 沪公网安备 31011502006855号
沪公网安备 31011502006855号